juuuding
[Computer Vision & Deep Learning] Image Processing - 이진 영상 본문
이진화
이진 영상이란 화소가 0 (흑) 혹은 1 (백)인 영상을 뜻한다. 이진 영상은 한 화소를 1 bit로 표현할 수 있지만 현대 컴퓨터는 메모리 용량이 크기 때문에 프로그램 편리성을 위해 1byte (8bit)를 사용한다. 컴퓨터 비전에서는 이러한 이진 영상을 "에지만 1로 표현하는 일" , "물체 검출 후 물체는 1, 배경은 0으로 표시하는 일" 등에 활용할 것이다.
명암 영상을 이진 영상으로 바꾸는 과정을 이진화라고 말한다. 이 때 임계값 T를 설정한 후 T보다 큰 화소는 1, 작은 화소는 0으로 바꾸어 이진화를 실행한다. 여기서 가장 중요한 것은 어떻게 이 임계값 T를 결정하느냐인데, 보통 히스토그램의 계곡 근처를 임계 값으로 결정한다. 여기서 말하는 히스토그램은 명암 단계를 가로축으로 두고, 이 명암 단계의 빈도수를 세로축으로 하여 그래프를 그린 것이다. 아래의 그림을 예시로 들자면 명암 단계 2와 6이 가장 보편적인 명암이 되고 계곡인 4가 임계값으로 설정된다.

하지만 실제 영상은 이것보다 크기도 크며, 명암 단계도 256이므로 계곡이 분명하지 않을 가능성이 더 크다. 아래는 그림 1의 2번 채널인 R채널에서 calcHist 함수를 이용하여 히스토그램을 구한 것이다.

import cv2 as cv
import sys
import matplotlib.pyplot as plt
img=cv.imread("resource/soccer.jpg")
if img is None:
sys.exit("파일을 찾을 수 없습니다")
h=cv.calcHist([img],[2],None,[256],[0,256])
plt.plot(h,color='g',linewidth=2)
plt.show()

※ calcHist 함수
calcHist 함수는 인수를 모두 리스트로 주어야 한다. 첫 번째 인수와 두 번째 인수는 영상과 영상의 채널 번호이다. 위의 코드에서는 [2]를 입력하였기 때문에 R 채널의 히스토그램을 구한다. 세 번째 인수는 히스토그램을 구할 영역을 지정하는 마스크이다. 여기서는 None을 입력했기 때문에 영상 전체에서 히스토그램을 구한다. 네 번째 인수는 히스토그램의 칸 수를 지정하는 것인데, 명암 단계가 256이기 때문에 256을 입력해주었다. 만약 여기에 128을 입력하면 0,1-> 0, 2,3 -> 1로 간주하여 128 칸의 히스토그램을 구한다. 마지막 인수는 세어볼 명암의 범위를 지정한다. 만약 [0,128]로 지정한다면 128 이상인 명암 값은 세지 않는다.
위의 히스토그램에서도 볼 수 있듯이 계곡이 하나로 분명하게 정해지지 않는다. 이러한 문제를 해결하고자 오츄 알고리즘을 적용해 보겠다.
오츄 알고리즘
오츄는 이진화를 식1의 최적화 문제로 바라보았다. 오츄 알고리즘에서는 모든 명암 값에 대해 목적 함수 J를 계산하고 J가 최소인 명암값을 최적값 t로 정한다. 그러고 t를 임계값으로 설정하여 영상을 이진화한다.
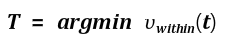
목적 함수 J(t)는 t의 좋은 정도를 측정하는 것인데 J가 작을 수록 좋다. 임의의 명암값 t로 이진화를 했을 때 0이 되는 화소의 분산과 1이 되는 화소의 분산의 가중치 합을 J로 설정하고 가중치는 해당 화소의 개수로 잡았다. 분산이 작을수록 0, 1 화소 집합이 균일하다는 의미이므로 좋은 이진 영상이라는 의미에서 생각해낸 방법이다.
오츄 알고리즘을 적용하여 이진화한 코드와 결과는 다음과 같다.
import cv2 as cv
import sys
img=cv.imread("resource/son.jpg")
t,bin_img=cv.threshold(img[:,:,2],0,255,cv.THRESH_BINARY+cv.THRESH_OTSU)
print("오츄 알고리즘의 최적 임계값 ",t)
cv.imshow("original",img)
cv.imshow("otsu",bin_img)
cv.imwrite("resource/otsu.jpg",bin_img)
cv.waitKey()
cv.destroyAllWindows()

참고로 threshold 함수의 첫 번째 인수는 영상의 부분과 채널을 지정, 두 번째 인수는 명암 값 범위를 지정해주는 것이다. 그리고 세 번째 인수에 cv.THRESH_BINARY+cv.THRESH_OTSU 을 작성하므로써 오츄 알고리즘을 적용하여 이진화를 수행한다. 위의 코드를 실행하면 최적 임계값 t가 115라는 사실도 알 수 있다.
연결 요소
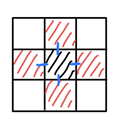
화소의 연결성에는 아래의 그림과 같이 4-연결성, 8-연결성이 있다. 4는 상하좌우만 중심 화소와 연결돼 있다고 여기고, 8은 상하좌우+대각선까지 중심화소와 연결돼 있다고 여기는 것이다.

이진 영상에서는 1의 값을 가진 연결된 화소의 집합을 연결 요소라고한다. 연결 요소는 connectecComponents 함수로 찾을 수 있다.
모폴로지
영상을 변환하는 과정에서 한 물체가 여러 영역으로 나누어 지거나, 여러 물체가 한 영역으로 붙는 경우가 발생한다. 이러한 현상을 최소화하기 위해 "모폴로지 연산"을 이용한다. 모폴로지는 구조 요소를 이용해 영역의 모양을 조작한다. 모폴로지의 기본 연산에는 팽창과 침식이 있다.
먼저 팽창은 구조 요소의 중심을 1인 화소로 설정한 다음 구조 요소에 해당하는 모든 화소를 1로 바꾸는 것이다. 다음으로 침식은 구조 요소의 중심을 1인 화소로 설정한 다음 구조 요소에 해당하는 화소 중 0인 화소가 하나라도 있다면, 해당 구조
요소에 포함되는 화소들을 모두 0으로 바꾸는 것이다.

이러한 방식을 적용한 팽창은 작은 홈을 메우고 끊어진 영역을 연결하는 역할을 하며, 침식은 돌출 부분을 깎는 역할을 한다. 참고로 한 영상을 침식한 후 팽창을 하면 열림, 팽창한 후 침식 하면 닫힘이라고 말한다. 다음은 모폴로지 영상을 적용한 코드이다.
※ png 파일은 채널이 4개이다.
import cv2 as cv
import numpy as np
import matplotlib.pyplot as plt
# png 파일로 한다면 채널 3을 유지해야하기 때문에 IMREAD_UNCHANGED 필요
#img=cv.imread("resource/son_sig.png",cv.IMREAD_UNCHANGED)
img=cv.imread("resource/son.jpg")
# 오츄 이진화를 이용하여 컬러 영상을 이진 영상으로 만들어줌
t,bin_img=cv.threshold(img[:,:,2],0,255,cv.THRESH_BINARY+cv.THRESH_OTSU)
plt.imshow(bin_img,cmap='gray') #cmap='gray'로 명암 영상으로 출력
plt.xticks([])
plt.yticks([])
plt.show()
b=bin_img[bin_img.shape[0]//4:bin_img.shape[0]*3//4,bin_img.shape[1]//4:bin_img.shape[1]*3//4] #영상의 가운데 부분만 자르기
plt.imshow(b,cmap='gray')
plt.xticks([])
plt.yticks([])
plt.show()
# 구조 요소
se=np.uint8([[0,0,1,0,0],[0,1,1,1,0],[1,1,1,1,1],[0,1,1,1,0],[0,0,1,0,0]])
# 팽창
b_dilation=cv.dilate(b,se,iterations=1)
plt.imshow(b_dilation,cmap='gray')
plt.xticks([])
plt.yticks([])
plt.show()
# 침식
b_erosion=cv.erode(b,se,iterations=1)
plt.imshow(b_erosion,cmap='gray')
plt.xticks([])
plt.yticks([])
plt.show()
# 팽창 후 침식 (닫기)
b_closing=cv.erode(cv.dilate(b,se,iterations=1),se,iterations=1)
plt.imshow(b_closing,cmap='gray')
plt.xticks([])
plt.yticks([])
plt.show()
# 침식 후 팽창 (열기)
b_opening=cv.dilate(cv.erode(b,se,iterations=1),se,iterations=1)
plt.imshow(b_opening,cmap='gray')
plt.xticks([])
plt.yticks([])
plt.show()
'인공지능 > cs231n' 카테고리의 다른 글
| [CS231n] Lecture 2. Image Classification (1) | 2023.11.28 |
|---|---|
| [CS231n] Lecture 1. Introduction and Historical Context (0) | 2023.11.23 |
| [Computer Vision & Deep Learning] Image Processing - 디지털 영상 기초 (1) | 2023.11.01 |
| [Computer Vision & Deep Learning] OpenCV - 페인팅 기능 만들기 (0) | 2023.10.31 |
| [Computer Vision & Deep Learning] OpenCV - 그래픽 기능과 사용자 인터페이스 만들기 (0) | 2023.10.31 |



